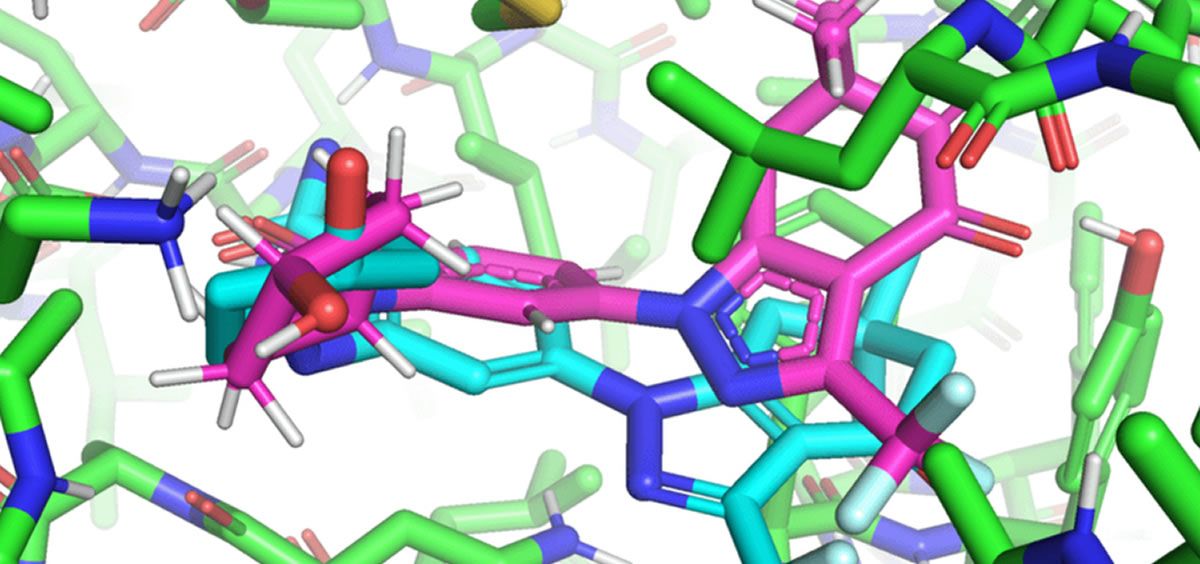
Los investigadores del MIT han desarrollado un modelo geométrico de aprendizaje profundo llamado EquiBind que es 1.200 veces más rápido en unir con éxito moléculas similares a fármacos que uno de los modelos computacionales de acoplamiento molecular más rápidos existentes, QuickVina2-W.
Antes de que pueda llevarse a cabo el desarrollo de fármacos, los investigadores deben encontrar moléculas prometedoras similares a fármacos que puedan unirse o "acoplarse" adecuadamente a ciertas proteínas objetivo en un proceso conocido como descubrimiento de fármacos. Después de acoplarse con éxito a la proteína, el fármaco de unión puede detener el funcionamiento de una proteína. Si esto le sucede a una proteína esencial de una bacteria, puede matar a la bacteria, confiriendo protección al cuerpo humano.
Sin embargo, el proceso de descubrimiento de fármacos puede ser costoso tanto desde el punto de vista financiero como informático. Además, el 90% de todos los medicamentos fallan una vez que se prueban en humanos debido a que no tienen efectos o tienen demasiados efectos secundarios. Una de las formas en que las compañías farmacéuticas recuperan los costes de estos fracasos es aumentando los precios de los medicamentos que tienen éxito.
Este modelo es más rápido y preciso que los modelos computacionales de última generación, lo que reduce las posibilidades y los costes de fallos en los ensayos de medicamentos
El proceso computacional actual para encontrar moléculas candidatas a fármacos prometedoras es así: la mayoría de los modelos computacionales de última generación se basan en un muestreo pesado de candidatos junto con métodos como puntuación, clasificación y ajuste fino para obtener el mejor "ajuste" entre los ligando y la proteína.
Hannes Stärk, estudiante de posgrado de primer año en el Departamento de Ingeniería Eléctrica y Ciencias de la Computación del MIT y autor principal del artículo, compara las metodologías típicas de unión de ligando a proteína con "tratar de encajar una llave en una cerradura con muchos agujeros".
A diferencia de la mayoría de los modelos que requieren varios intentos para encontrar una posición favorable para el ligando en la proteína, EquiBind ya tiene un razonamiento geométrico incorporado que ayuda al modelo a aprender la física subyacente de las moléculas y a generalizar con éxito para hacer mejores predicciones al encontrar nuevos datos no vistos. .
La publicación de estos hallazgos atrajo rápidamente la atención de los profesionales de la industria, incluido Pat Walters, director de datos de Relay Therapeutics. Walters sugirió que el equipo pruebe su modelo en un fármaco y una proteína ya existentes que se usan para el cáncer de pulmón, la leucemia y los tumores gastrointestinales. Mientras que la mayoría de los métodos de acoplamiento tradicionales no pudieron unir con éxito los ligandos que funcionaron en esas proteínas, EquiBind tuvo éxito.