Un equipo de investigadores del Consejo Superior de Investigaciones Científicas (CSIC) ha desvelado nuevos datos sobre el mecanismo de resistencia en Pseudomonas, una bacteria multiresistente, con gran incidencia en hospitales y responsable de infecciones pulmonares y respiratorias, entre otras.
Según explican desde el Consejo, este estudio, publicado en la revista PNAS, abre la puerta al desarrollo de nuevos antimicrobianos que reduzcan la resistencia en este tipo de patógenos multirresistentes.
Según el director del trabajo, Juan A. Hermoso, investigador del CSIC en el Instituto de Química-Física Rocasolano, “los antibióticos usados más ampliamente (los beta-lactámicos, como la penicilina) combaten las bacterias bloqueando las maquinarias celulares que sintetizan la pared bacteriana, que es clave en su supervivencia”. A su vez, este experto detalla que “esta acción contra las maquinarias celulares provoca que las fibras que componen esa pared no se unan entre sí y adquieran formas aberrantes, que conducen a la muerte de la bacteria. Sin embargo, la bacteria Pseudomonas ha desarrollado un mecanismo para resistir a la acción del antibiótico”
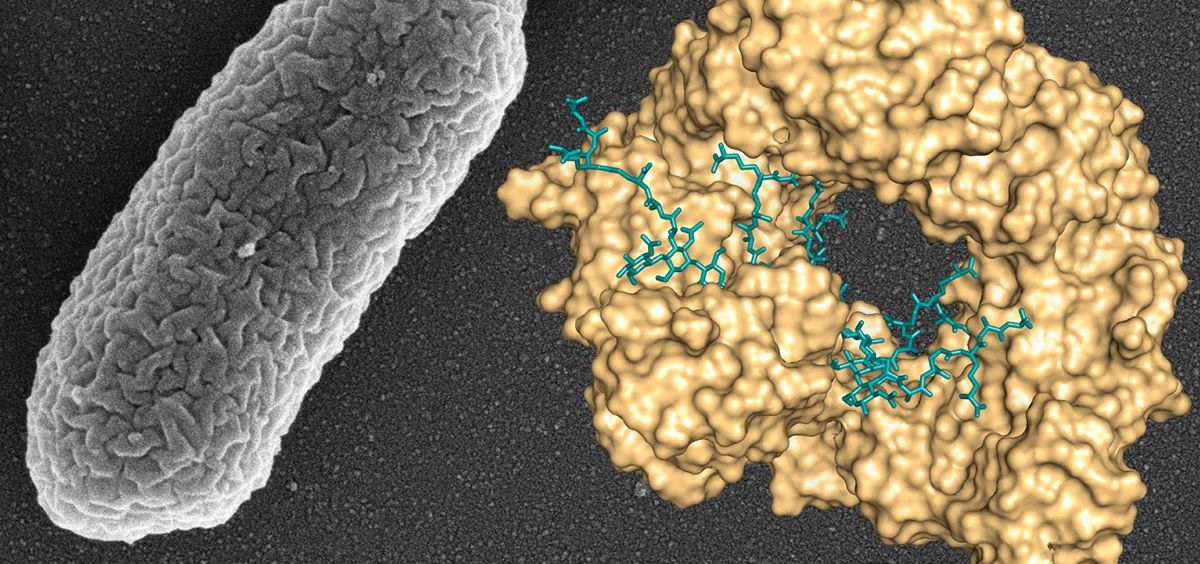
“Mediante cristalografía de rayos X se han determinado las estructuras tridimensionales de la enzima Slt, en complejo con distintos fragmentos de la pared bacteriana, lo que ha permitido diseccionar el mecanismo por el cual esta enzima contribuye a la resistencia de Pseudomonas”
Siguiendo esta línea, el equipo de investigación de Juan A. Hermoso ha descubierto que “mediante cristalografía de rayos X se han determinado las estructuras tridimensionales de la enzima Slt, en complejo con distintos fragmentos de la pared bacteriana, lo que ha permitido diseccionar el mecanismo por el cual esta enzima contribuye a la resistencia de Pseudomonas”, apunta el director del estudio.
De este modo, “cuando el antibiótico bloquea la biosíntesis de la pared bacteriana, la enzima Slt es capaz de unirse a esas fibras, mediante un mecanismo hasta ahora desconocido, y empieza a cortarlas progresivamente en fragmentos más pequeños”, ha señalado Hermoso.
A su vez, desde el CSIC continúan que estos fragmentos son internalizados, “donde son reconocidos por un receptor que inicia la producción de una beta-lactamasa (enzima que degrada antibióticos beta-lactámicos) que, una vez exportada al exterior, eliminará los antibióticos de la pared bacteriana”. Así, el mecanismo de reconocimiento y ruptura de la pared por Slt desencadena la respuesta de resistencia a los antibióticos beta-lactámicos en Pseudomonas.